De 2019 à 2021, 5000 heures de travail ont été consacrées par l’équipe d’ELISOL au projet Menninov (« Méthodes Innovantes en Nématologie »), un projet financé dans le cadre de READYNOV (région Occitanie), porté par ELISOL et réalisé en collaboration avec l’IRD.
Le projet MENINNOV a cherché à développer 2 nouvelles méthodes : une basée sur des techniques moléculaires et l’autre sur l’analyse d’image pour réaliser des opérations actuellement basées sur l’observation microscopique et tester l’interopérabilité des différents types de méthodes en terme de robustesse des résultats. Ce projet a été réalisé en collaboration S. Bellafiore, chercheur de l’Institut de Recherche pour le Développement (IRD) qui s’intéresse tout particulièrement à l’étude des interactions entre les nématodes phytoparasites et les plantes.
Les outils d’évaluation de la qualité des sols (communautés de nématodes) et de l’état sanitaire des sols (nématodes phytoparasites) sont très chronophages, c’est ce qui limite actuellement le débit analytique : temps nécessaire pour former des experts en taxonomie en amont et temps de réalisation des analyses nématologiques.
Deux tâches distinctes ont été conduites dans le cadre de ce projet.
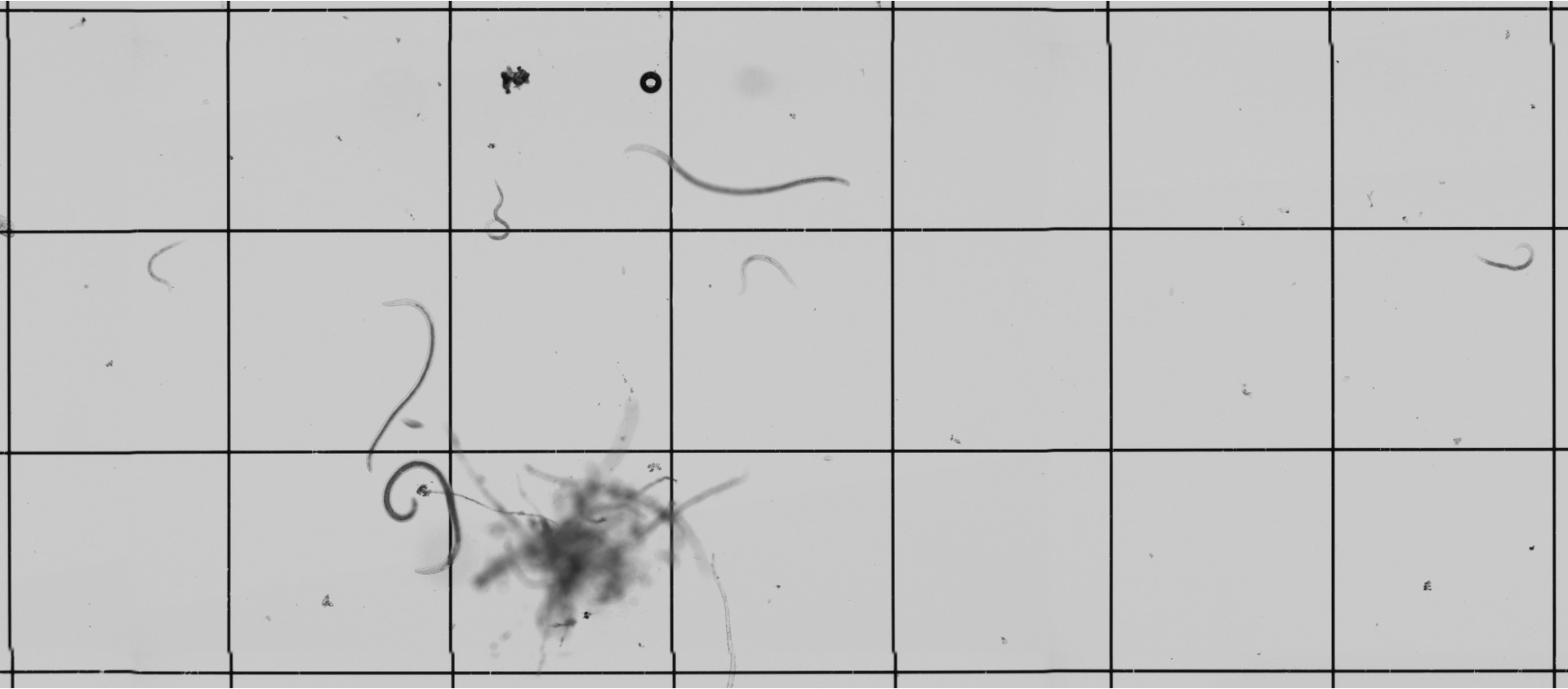
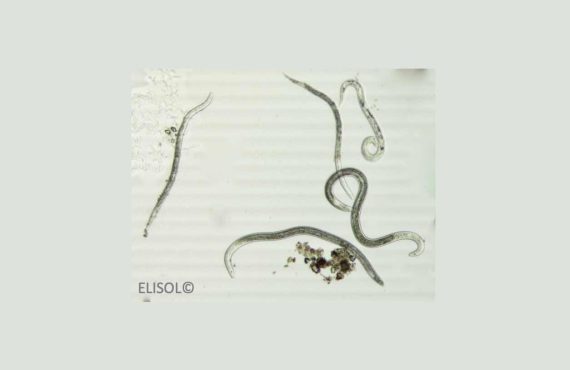
Tâche 1 : Développement d’une méthode automatisée de dénombrement des nématodes dans un échantillon, comptage par analyse d’image (recherche interne ELISOL) :
Cette opération a donné de très bons résultats en utilisant une méthode de « machin learning » et des réseaux de neurones à convolutions.
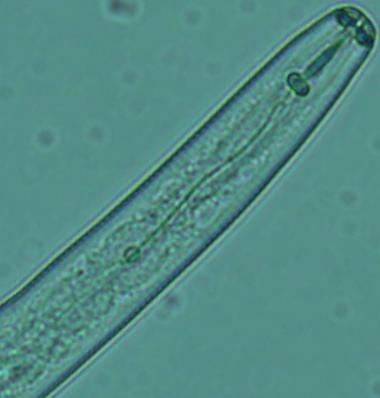
Le dénombrement de population de nématodes C. elegans colorés est réalisé automatiquement avec un taux d’erreur inférieur à 5%. La méthode reste à affiner pour obtenir des résultats aussi satisfaisants avec des communautés de nématodes complexes issues d’échantillons de sol. Un nouveau projet démarre en 2022 pour finaliser cette méthode.
Tâche 2 : Développement d’une méthode de caractérisation des communautés de nématodes du sol par biologie moléculaire, et plus précisement par amplicon-barcoding (recherche en collaboration ELISOL & IRD) :
Le principe a été de travailler sur les nématodes préalablement extraits du sol par élutriation Oostenbrink afin d’avoir accès à la diversité totale des nématodes : cette méthode étant la plus exhaustive pour obtenir les nématodes du sol.
Tout d’abord, les différentes étapes nécessaires au pipe-line de biologie moléculaire ont été mises au point :
1) procédé de fixation des nématodes permettant la conservation de leur ADN, 2) extraction de l’ADN des nématodes, 3) sélection des amorces correspondant au fragment d’intérêt retenu, 4) mise au point des PCR pour obtention des amplicons, 5) pipe-line de bio-informatique d’analyse des séquences dont l’obtention a été sous-traitée.
Puis, la démarche a été réalisée avec 100 échantillons :
– Sélection de 100 échantillons de sol de provenance géographique et d’usages du sol variés ;
– Extraction des nématodes du sol par la méthode classique d’analyse de la nématofaune ;
– Dénombrement, pour chaque échantillon de sol, des nématodes totaux (abondance totale) puis séparation en 2 lots : pour analyse moléculaire (lot1) et analyse morphologique (lot2) ;
– Analyse moléculaire sur le lot 1 (en suivant les étapes mises au point préalablement) ;
– Analyse morphologique des nématodes sur le lot 2 ;
– Analyse croisée des résultats de l’analyse moléculaire et de l’analyse morphologique.
L’objectif du projet a été atteint : la nématofaune de 100 échantillons d’origine variée (96 échantillons en fait pour être exact !) a pu être comparée entre analyse morphologique et analyse moléculaire. ELISOL environnement s’est même doté en 2021 d’un laboratoire de biologie moléculaire lui permettant d’être autonome dans ces analyses.
En ce qui concerne la comparaison des résultats entre les deux méthodes, il y aurait beaucoup à dire ! Il y a des aspects positifs mais plusieurs points faibles rendent la méthode moléculaire testée encore insuffisamment précise à ce jour pour être adoptée. Un point faible majeur réside dans le fait que les « nombre de reads » ne peuvent pas être corrélés aux abondances de nématodes (nombre d’individus par gramme de sol). Par ailleurs, en ne travaillant que sur 1 seul fragment d’ADN (de l’ordre de 300 pb), il n’est pas possible de discriminer tous les taxons avec un niveau de précision suffisant (= au moins regroupement d’espèces appartenant à la même guilde fonctionnelle) ; pour obtenir des regroupements exploitables du point de vue écologique, il serait nécessaire de faire l’analyse sur 2 ou 3 fragments simultanément.
La région Occitanie a soutenu ce projet et l’a financé dans le cadre de l’appel à projet MENINNOV. Un grand merci à elle!